综合新闻
博士研究生刘博等在《Nucleic Acids Research》杂志发表研究论文介绍病原菌毒力因子分析系统VFanalyzer
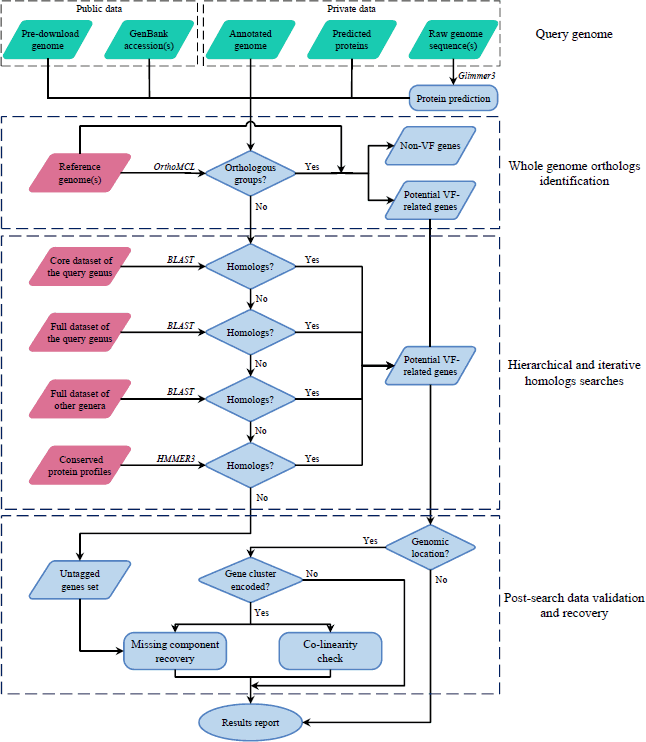
近年来新兴的第三代测序技术(如Pacific Biosciences和Oxford Nanopore)能够产生出几Kbp甚至几十Kbp的长序列片段,因而使得从头测序(de novo sequencing)逐渐取代重测序(resequencing)成为病原菌基因组学研究的重要技术。由此产生的大量病原菌完整基因组序列,促使研究人员对病原菌毒力因子的快速准确的数据分析产生了迫切的需求。病原菌毒力因子数据库(简称VFDB,http://www.mgc.ac.cn/VFs/)由我所重点实验室2004年建立并长期维护,经过十多年的持续升级和更新已经发展成为国际上最大的病原菌毒力因子资源中心,被国内外同行广泛使用。在此基础上,杨剑课题组的博士研究生刘博等自主设计开发了全新的病原菌毒力因子在线分析系统VFanalyzer。该系统首先通过直系同源蛋白(orthologs)的识别避免了由旁系同源(paralogs)带来的假阳性。然后利用VFDB的分层数据集进行循环迭代式的相似性检索,从而显著提高了毒力因子识别的灵敏度和特异性。最后,还设计开发了基于基因簇位置信息的毒力因子识别优化算法,使得自动分析系统VFanalyzer达到了毒力因子识别准确率接近人工分析的水平。该分析系统一经推出,立刻受到了国内外相关领域研究人员的广泛关注。截至目前,系统在线运行不到四个月,已经完成了来自全球的超过一千株病原菌基因组的毒力因子分析任务。近日,VFanalyzer相关研究论文在线发表于生物信息学领域国际著名期刊《Nucleic Acids Research》2019年数据库专刊。